



Fare i LEGO® con il DNA
(English version - Android app version)
(una pagina web didattica che dimostra alcuni concetti delle nanotecnologie basate sul DNA ... e vi ci fa giocare un po')
copyright: Giampaolo Zuccheri 2021
Il DNA è la molecola alla base del codice della vita e si ritrova chimicamente uguale in tutti gli organismi. La chimica e la struttura del DNA sono perfette per codificare quelle informazioni che possono portano al legame specifico tra due o più molecole. La vita stessa dipende dall'efficacia e dalla specificità di questi legami.
Ogni molecola di DNA è caratterizzata dalla sua sequenza: è come una parola che descrive la sua struttura chimica. Le sequenze di due molecole di DNA (o due tratti di molecole) si dicono complementari quando il codice informativo che portano può mediare la loro interazione specifica (cioè fa sì che si possano legare tra loro). Il codice della complementarità del DNA è stato scoperto da James Watson e Francis Crick. Sfruttando la conoscenza di questo codice informativo, assimilabile ad una serie di regole grammaticali, possiamo progettare la sequenza di molecole di DNA sintetiche in modo che si leghino tra loro. Queste molecole sono create in laboratorio e non hanno necessariamente alcuna funzione biologica, ma possono permettere di costruire dei "mattoncini" e poi di montare tra loro mattoncini diversi per fare costruzioni via via più complesse, in modo simile a quello che possiamo fare con i LEGO®. La differenza tra le nanotecnologie basate sul DNA e i LEGO® "normali" è che i mattoncini e le costruzioni risultanti sono estremamente piccole (nella scala dimensionale dei nanometri) e che non servono le mani per costruire i mattoncini o per assemblare le costruzioni. Tutto avviene automaticamente: basta progettare le sequenze giuste, sintetizzarle e scioglierle tutte assieme in una soluzione acquosa.
A cosa servono i LEGO® di DNA (che noi a volte chiamiamo semplicemente nanostrutture di DNA)? Per ora non ci sono tante applicazioni pratiche, ma ci sono dimostrazioni che con il controllo del loro assemblaggio, della loro struttura e del loro comportamento si potranno costruire materiali innovativi, produrre catalizzatori più efficienti (utili per inquinare di meno) o anche farmaci innovativi.
 |
Una molecola di DNA è una catena flessibile, paragonabile ad uno spaghetto cotto (ma molto più sottile). In una soluzione, le molecole sono molto numerose e nuotano con direzioni casuali, a volte incontrandosi. Se due molecole di DNA con sequenze qualsiasi si incontrano, normalmente non interagiscono, come mostrato nella simulazione di sotto (i diversi colori rappresentano diverse sequenze). |  |
(schiacciate la bandierina verde per riavviare le simulazioni o il cartello STOP per fermarla).
 |
Ma se invece le due molecole hanno sequenze complementari tra loro e per caso si incontrano, si legano in modo specifico. Legandosi, inoltre, le due molecole flessibili "a spaghetto cotto" formano una doppia elica più rigida (forse l'avrete vista rappresentata anche nei libri di scuola o su altri siti web). Nelle nostre rappresentazioni grafiche, le sequenze scure sono complementari a quelle chiare dello stesso colore. Guardate nella simulazione sotto cosa succede quando due molecole di DNA complementari per caso si incontrano. In una soluzione, possono essere presenti tante molecole di DNA con sequenze diverse: solo quelle complementari incontrandosi si legano, le altre non interagiscono. |
(schiacciate la bandierina verde per riavviare le simulazioni o il cartello STOP per fermarla).
Per semplicità, ai fini di questa dimostrazione, possiamo identificare un tratto di sequenza di DNA (una 'parola') con una lettera, ad esempio "A" o "a". In questa simbologia, consideriamo "A" complementare ad "a", "B" complementare a "b", e così via (lettera maiuscola complementare a lettera minuscola). Una molecola di DNA (uno 'spaghetto') può avere una sequenza costituita da un solo tratto o da più tratti in fila (che potremmo rappresentare, ad esempio, con "AcB" che mostra che sono tratti diversi). Le molecole di DNA hanno caratteristiche chimiche che fanno sì che si leghino tra loro in modo 'antiparallelo,' come se la testa di una sequenza di legasse ai piedi della sua sequenza complementare, e viceversa. Graficamente si rappresentano le sequenze come frecce con una punta: nelle doppie eliche formate da due sequenze complementari le punte delle due frecce sono orientate in maniera opposta, come nel disegno quì a fianco. I 'bastoncini' che collegano due molecole di DNA sono i legami chimici che si instaurano tra due molecole complementari per tenerle unite in un'elica. |
 |
Se una molecola è formata da più tratti di sequenze in fila, si dovrà tenere conto dell'antiparallelismo per progettare la sua sequenza complementare (e costruire il primo mattoncino LEGO®). Ad esempio, "AB" si legherà (è complementare) a "ba" e non ad "ab" o "AB".
Progettate ora il vostro primo mattoncino: dovete ottenere 2 sequenze complementari, scegliendo tra le opzioni con i selettori. Poi verificate se la vostra risposta è esatta quando le molecole si incontrano nella simulazione. La struttura che volete ottenere è questa:

e la possiamo schematizzare così:
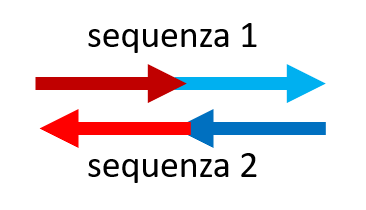
(schiacciate la bandierina verde per riavviare le simulazioni o il cartello STOP per fermarla. Ricaricate la pagina web per azzerare eventuali vostre scelte).
Per fare i LEGO® di DNA, spesso si usano strutture ramificate, come le giunzioni a 3 o 4 braccia. In questo modo si possono ottenere tante forme diverse. Questi mattoncini sono fatti progettando le sequenze di 3 o 4 pezzi diversi, ciascuno costituito da 2 tratti complementari tra loro a 2 a 2 (come hai fatto prima).
Prova ora a progettare le sequenze (scegliendo le opzioni con i selettori) per costruire questa giunzione a 3 braccia:

schematizzabile così:

(schiacciate la bandierina verde per riavviare le simulazioni o il cartello STOP per fermarla. Ricaricate la pagina web per azzerare eventuali vostre scelte).
A questo punto sapete come costruire alcuni tipi di mattoncini di DNA (ce ne sono tanti diversi). Il divertimento nei LEGO® è attaccare tra loro i mattoncini. Lo si fa anche con i mattoncini di DNA! Bisogna dotare i mattoncini di DNA di "code coesive" che leghino in modo specifico e automatico un mattoncino all'altro.
Le code coesive sono brevi tratti di sequenze tra loro complementari: quando si incontrano fanno delle doppie eliche a ponte tra due mattoncini e così li montano automaticamente.
Ora vi invitiamo a progettare le code (i tratti viola nello schema) per il montaggio di 3 mattoncini di DNA. Dovete ottenere la struttura rappresentata quì sotto. Scegliete le code giuste con i selettori.

che si schematizza come:
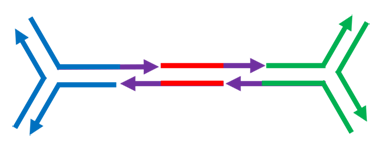
(schiacciate la bandierina verde per riavviare le simulazioni o il cartello STOP per fermarla. Ricaricate la pagina web per azzerare eventuali vostre scelte).
Congratulazioni! siete giunti alla fine di questa dimostrazione sulle nanotecnologie basate sul DNA.
Se avete commenti o siete interessati ad approfondire l'argomento, scrivete a Giampaolo Zuccheri.
Nel video di sotto, una descrizione delle nostre attività sull'argomento:
